
Stuhlproben für Mikrobiom-Analysen .
Die OMNIgene®-GUT Kits OM-200 und OMR-205 erfüllen alle Voraussetzungen für beste Probenqualität und ein realitätsnahes Mikrobiomprofil: Die Probennahme selbst ist einfach und sauber, perfekt für Eigenbeprobung durch Patienten oder Studienteilnehmer. Die Stuhlprobe wird im Röhrchen sofort homogenisiert, die Konservierungslösung stabilisiert Nukleinsäuren sehr effektiv. Diese genetische Momentaufnahme der Mikrobiota bleibt auch ohne Kühlung während Lagerung und Transport längere Zeit erhalten.
Hard Facts .
- Intuitive und benutzerfreundliche Probengewinnung
- Minimierung von mikrobiellem Wachstum
- Minimierung von DNA- und RNA-Degradation
- Stabilisierung der DNA und RNA bei Temperaturschwankungen (-20°C bis 50°C)
- Sehr gute Signal-To-Noise Ratio in nachfolgenden Datenanalysen

Anwendungen .
Zur Minimierung der Verfälschung des Darm-Mikrobioms ist eine perfekte Probenhomogenisierung und -stabilisierung notwendig. Aus den gewonnenen Proben können verschiedenste Mikroben zuverlässig über Downstream-Applikationen nachgewiesen werden.
Macklaim, J., Tayeb, M., Dillane, C., Fragel, C., & Cunningham, L. (2021). The need for stabilization: Short-term variation in microbiome profiles. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00070.pdf
Doukhanine, E., Bouevitch, A., Pozza, L., & Merino, C. (2014). OMNIgene®•GUT enables reliable collection of high quality fecal samples for gut microbiome studies. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00040.pdf
Doukhanine, E., Bouevitch, A., Brown, A., Gage LaVecchia, J., Merino, C., & Pozza, L. (2016). OMNIgene®•GUT stabilizes the microbiome profile at ambient temperature for 60 days and during transport. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00042.pdf
Brown, A., Lynch, D., Bouevitch, A., & Doukhanine, E. (2018). OMNIgene®•GUT provides easy self-collection and stabilization of liquid fecal samples for microbiome profiling. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00056.pdf
Tayeb, M., Macklaim, J., Colemeco, S., Fragel, C., Dillane, C., & Cunningham, L. (2020). Impact of fecal collection methods on sample homogenization and extraction reproducibility in gut microbiome profiling. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00064.pdf
16S rRNA-Analysen ermöglichen die Detektion, Identifizierung und Charakterisierung der Mikroorganismen des Darmmikrobioms.
Anderson, E. L., Li, W., Klitgord, N., Highlander, S. K., Dayrit, M., Seguritan, V., Yooseph, S., Biggs, W., Venter, J. C., Nelson, K. E., & Jones, M. B. (2016). A robust ambient temperature collection and stabilization strategy: Enabling worldwide functional studies of the human microbiome. Scientific Reports, 6(1), 31731. https://doi.org/10.1038/srep31731
Choo, J. M., Leong, L. E., & Rogers, G. B. (2015). Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports, 5(1), 16350. https://doi.org/10.1038/srep16350
Das humane Darm-Mikrobiom rückt immer mehr in den Fokus der Forschung. Es mehren sich die Hinweise, dass das Mikrobiom in vielen physiologischen, pharmakokinetischen und pathologischen Prozessen eine ausschlaggebende Rolle spielt.
Abrahamson, M., Hooker, E., Ajami, N. J., Petrosino, J. F., & Orwoll, E. S. (2017). Successful collection of stool samples for microbiome analyses from a large community-based population of elderly men. Contemporary Clinical Trials Communications, 7, 158–162. https://doi.org/10.1016/j.conctc.2017.07.002
OMNIgene GUT Mikrobiom Kits .
Produktübersicht .
| Eigenschaft | OM-200 | OMR-205 |
|---|---|---|
| Abnahmestelle | Stuhl | Stuhl |
| Homogenisierung von Proben | ✔ | ✔ |
| Mikrobielle Nukleinsäuren | DNA | DNA und RNA |
| Entnommene Probe pro Kit (Median) | 520 ± 101 mg | 545 mg |
| Versand bei Umgebungstemperatur | ✔ | ✔ |
| Kompatibel mit einer Vielzahl von Nukleinsäureextraktions- und -aufreinigungsverfahren | ✔ | ✔ |
| Standardisiertes Format für die Verarbeitung mit hohem Durchsatz | ✔ | ✔ |
| Anzahl der Extraktionen pro Kit (250 µL pro Extraktion) | 8 | 10 (200 µl pro Extraktion) |
| DNA-Ausbeute pro Extraktion ⌀ (Median) | 12 ± 1,49 µg (12,56 µg) | 4,40 ± 0,52 µg (2,06 µg) |
| DNA mit hohem Molekulargewicht | ✔ | ✔ |
| Stabilität der Mikrobiom-DNA bei Raumtemperatur | 60 Tage | 30 Tage |
| Stabilität der Mikrobiom-RNA bei Raumtemperatur | – | 10 Tage |
| Geeignet für NGS-Downstream-Anwendungen | ✔ | ✔ |
| Geeignet für Langzeitlagerung und Biobanking | ✔ | ✔ |

OM-200 - lange DNA Stabilität .

OMNIgene® GUT ist ein All-in-One-System zur einfachen Selbstsammlung von Stuhlproben für die Erstellung von Darmmikrobiom-Profilen für Forschungszwecke.

- Verbesserte Compliance der Spender durch die intuitive und benutzerfreundliche Handhabung
- Einfache Eigenprobennahme durch Patienten und Studienteilnehmer
- Minimierung der durch mikrobielles Wachstum und DNA-Abbau verursachten Verzerrungen
- Eliminierung der Notwendigkeit des Wiegens und der Aliquotierung mit einer volumetrischen Standardprobe
- Die DNA bleibt im Probenröhrchen über 60 Tage stabil - keine Kühlkette erforderlich
- Aufrechterhaltung der DNA-Integrität bei Schwankungen der Umgebungstemperatur (z. B. -20 °C bis 50 °C)
- Sofortiges Homogenisieren und Stabilisieren der Probe bei der Entnahme
- Röhrchenformat eignet sich für die manuelle oder automatisierte Verarbeitung
- Qualitativ hochwertige DNA für 16S rRNA-Profiling, Shotgun Sequencing, qPCR, Arrays
- Barcodierung für vollständige Rückverfolgbarkeit der Proben
Weitere Informationen .
-
HerstellerwebseiteHerstellerwebseite
-
Gebrauchsanweisung (engl.)Gebrauchsanweisung (engl.)
OMR-205 - hohe DNA und RNA Stabilität .

OMNIgene® GUT OMR-205 ist ein All-in-One-System zur einfachen Selbstsammlung von Stuhlproben für die Stabilisierung von DNA und RNA zur Erstellung von Darmmikrobiom-Profilen für Forschungszwecke.

- Verbesserte Compliance der Spender durch die intuitive und benutzerfreundliche Handhabung
- Minimierung von Mikrobiom-Verzerrungen
- Stabilität der DNA 30 Tage und RNA 10 Tage bei Raumtemperatur
- Eliminierung der Notwendigkeit des Wiegens und der Aliquotierung mit einer volumetrischen Standardprobe
- Aufrechterhaltung der DNA- und RNA-Integrität bei Schwankungen der Umgebungstemperatur (z. B. -20 °C bis 50 °C)
- Sofortiges Homogenisieren und Stabilisieren der Probe bei der Entnahme
- Qualitativ hochwertige DNA und RNA für Multi-Omics Downstream Analysen
- Röhrchenformat eignet sich für die manuelle oder automatisierte Verarbeitung
- Barcodierung für vollständige Rückverfolgbarkeit der Proben
Weitere Informationen .
-
HerstellerwebseiteHerstellerwebseite
-
Gebrauchsanweisung (engl.)Gebrauchsanweisung (engl.)
All-in-One Versandkit OM-200+ACP102 .

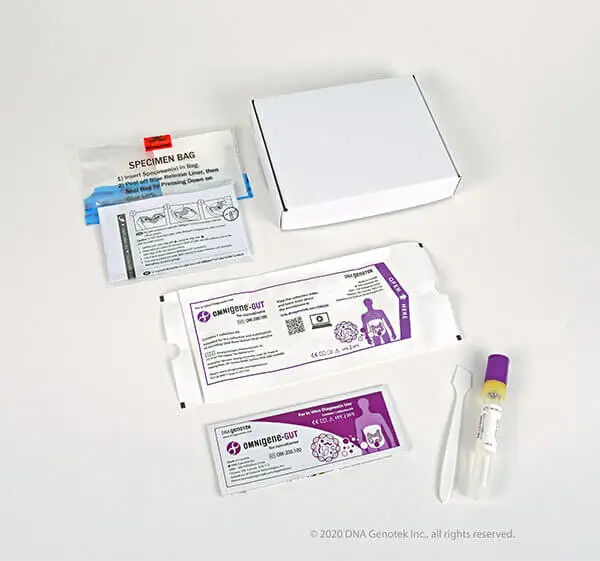
Konfiguration des Kits .
- OM-200: OMNIgene•GUT inklusive Benutzeranweisung und Spatel
- OM-AC1: Toilettenzubehör zur vereinfachten Probennahme
- MO-5: Bioproben-Beutel
- 2-Wege-Versandkarton (mit abziehbarem Klebestreifen für Probenrücksendung)
- Individuelles Label für Rückverfolgbarkeit
DNA Stabilität .
OMNIgene-GUT-stabilisierte Proben wurden zu Beginn (T0) und nach 14- bzw. 60-tägiger Lagerung bei Raumtemperatur (23°C) extrahiert. Gepaarte nicht stabilisierte Proben wurden zu Beginn (T0, frisch) und nach 14 Tagen bei Raumtemperatur (23°C) oder nach 14 Tagen bei -80°C extrahiert. Die Ähnlichkeit der Proben bei Ausgangswert und nach Lagerung wurde anhand der Bray-Curtis-Distanzen bewertet.
Aus: Doukhanine, E., Bouevitch, A., Brown, A., Gage LaVecchia, J., Merino, C., & Pozza, L. (2016). OMNIgene®•GUT stabilizes the microbiome profile at ambient temperature for 60 days and during transport. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00042.pdf
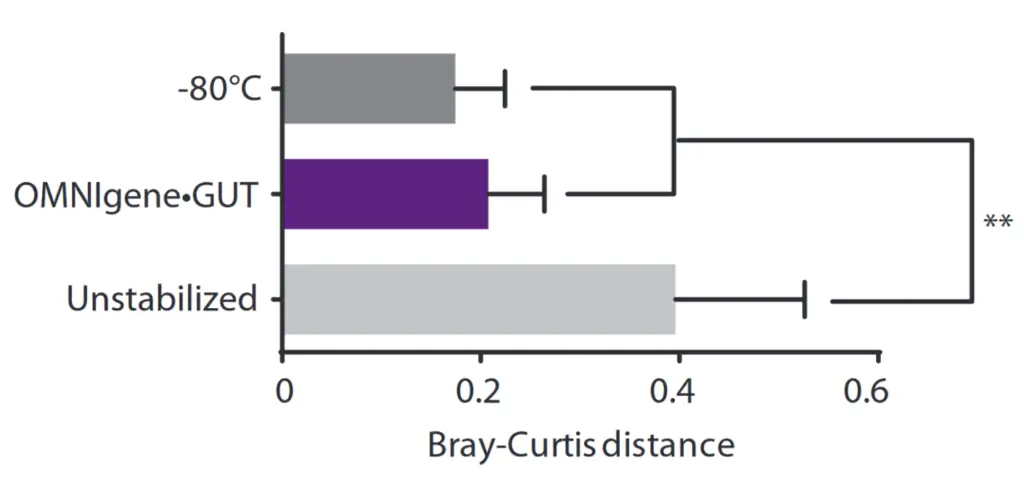
Bray-Curtis-Abstandsdifferenz zwischen unstabilisierten (14 Tage bei 23°C), OMNIgene-GUT (60 Tage bei 23°C) und -80°C (14 Tage bei -80°C) Proben im Vergleich zu frischen Proben. Signifikante Unähnlichkeit wurde mit Mann-Whitney (**P≤0,001) bewertet.
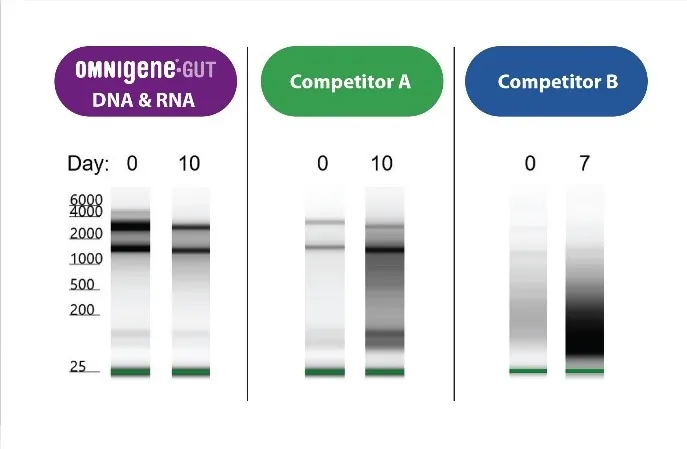
RNA Stabilität in OMR-205 .

© DNA Genotek, Inc. All rights reserved.
Keine Verzerrung in der qPCR .
Die Kompatibilität der mit OMNIgene-GUT gesammelten Proben mit dem PowerFecal DNA Isolation Kit wurde geprüft. Eine frische Probe sowie eine OMNIgene-GUT-Probe wurden aus derselben fäkalen Sammelprobe von drei verschiedenen Spendern gesammelt. Die DNA wurde aus einem Aliquot jeder Probe extrahiert und die DNA-Performance durch 16S rRNA qPCR bestimmt.
Aus: Doukhanine, E., Bouevitch, A., Pozza, L., & Merino, C. (2014). OMNIgene®•GUT enables reliable collection of high quality fecal samples for gut microbiome studies. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00040.pdf
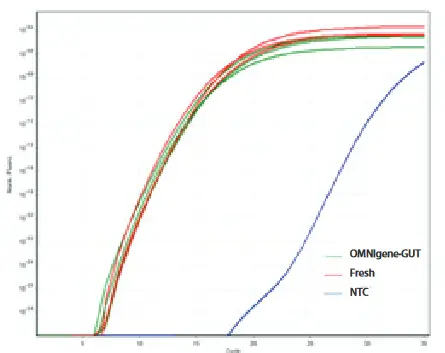
Realtime 16S rRNA PCR von fäkaler DNA, die von von 3 Spendern unter Verwendung von OMNIgene-GUT oder frischen Proben extrahiert wurden (∆∆Cq = 0,54). NTC – no-Template Kontrolle.
Kein Shift des Mikrobiomprofils während der Lagerung .
Brown, A., Lynch, D., Bouevitch, A., & Doukhanine, E. (2018). OMNIgene®•GUT provides easy self-collection and stabilization of liquid fecal samples for microbiome profiling. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00056.pdf
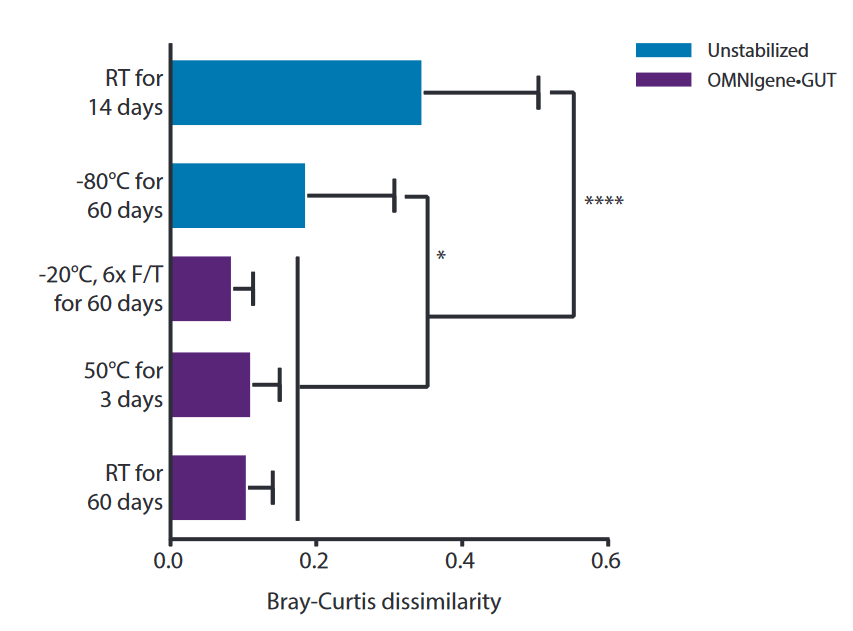
Bray-Curtis-Dissimilarität von Stuhlproben, die mit OMNIgene-GUT von Spendern mit Dysbiose gesammelt und dann Lagerung und simulierten Versandbedingungen ausgesetzt waren. Die Dissimilarität wurde durch den Vergleich des mikrobiellen Profils der Probe zu Beginn der Untersuchung mit gepaarten Proben ermittelt. Ein signifikanter Unterschied im Mikrobiomprofil wurde bei unstabilisierten (blau) Proben beobachtet, die bei bei RT oder -80°C aufbewahrt wurden, im Vergleich zu OMNIgene-GUT (lila) stabilisierten Proben (*P≤0,05 bzw. ****P≤0,0005).
Kein Shift des Mikrobiomprofils währende der Lagerung .
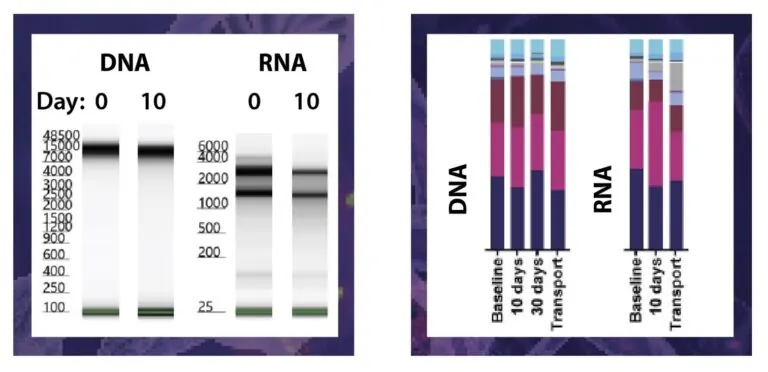
OMNIgene-GUT Proben wurden mit OMR-205 10 Tage lang bei Raumtemperatur gelagert und simulierten Transportbedingungen ausgesetzt. Die Verzerrung des Mikrobiom-Profils (DNA und RNA) ist im Vergleich zu frisch entnommenen Proben minimal.
Realistischer Snapshot des Mikrobioms .
Die mit OMNIgene Kits gewonnen Proben sind von hervorragender Qualität. Die hohe Integrität der stabilisierten DNA ermöglicht eine sehr realistische Darstellung des Mikrobiom-Profils.
Aus: DNA Genotek. (n.d.). OMNIgene Devices.
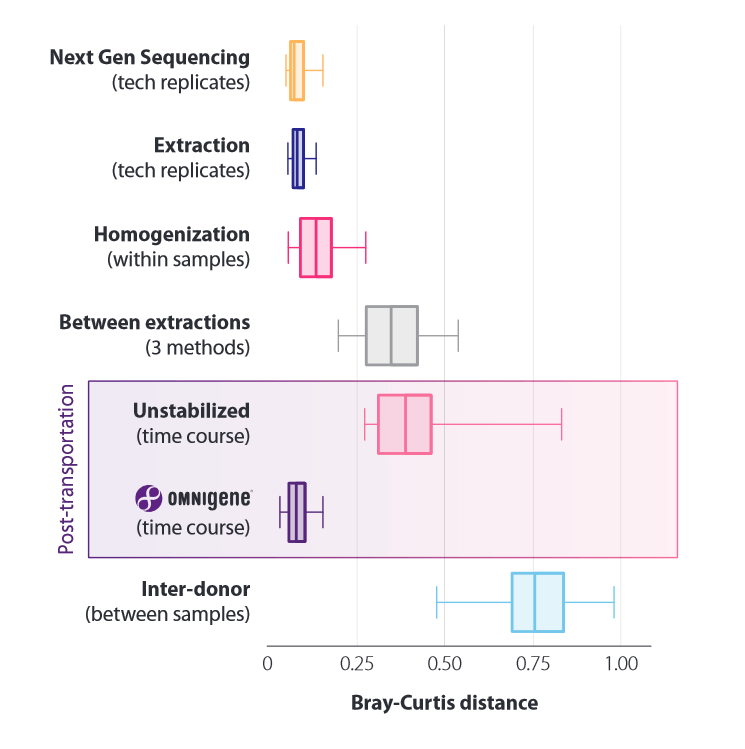
Quantifizierung der Auswirkungen von präanalytischen Faktoren: Mikrobiom-Profilunterschiede zwischen technischen Replikaten bei der Sequenzierung, der Extraktion, der Probenhomogenisierung, von 3 verschiedenen Kits im Vergleich. Analyse erfolgte im Hinblick auf Transport (Temperaturschwankungen zwischen -8°C und +23°C, unstabilisiert und OMNIgene) und Spender zu Spender (Kontrolle).
Hohe Stabilität des Mikrobiomprofils .
Während des Transports von der Probennahme zum verarbeitenden Labor sind die Proben in der Regel ungünstigen Bedingungen ausgesetzt. Um diese zu simulieren, wurden unstabilisierte und mit OMNIgene-GUT stabilisierte Proben drei Tage lang bei 50°C oder 37°C gelagert oder mehreren Gefrier-Auftau-Zyklen ausgesetzt.
Aus: Doukhanine, E., Bouevitch, A., Brown, A., Gage LaVecchia, J., Merino, C., & Pozza, L. (2016). OMNIgene®•GUT stabilizes the microbiome profile at ambient temperature for 60 days and during transport. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00042.pdf
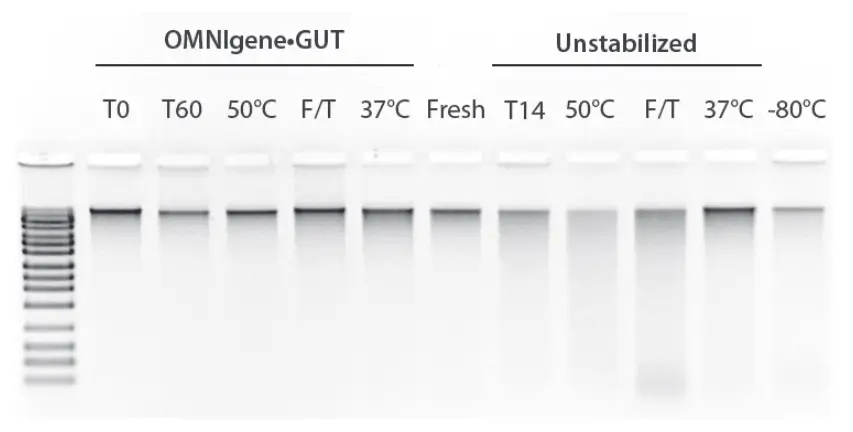
DNA-Integrität von OMNIgene-GUT-Proben unter simulierten Transportbedingungen. OMNIgene-GUT Proben von einem repräsentativen Spender wurden 60 Tage lang bei 23°C (T60), 50°C für 3 Tage (50°C) oder 37°C für 3 Tage (37°C) gelagert; oder 6 Gefrier-Auftau-Zyklen (F/T) unterzogen. Unstabilisierte Proben desselben Spenders wurden 14 Tage bei 23°C (T14), 1 Tag bei 50°C (50°C) oder 3 Tage bei 37°C (37°C) oder bei 14 Tage bei -80°C (-80°C) gelagert.

