
Probennahme für Haut-Mikrobiom .
OMNIgene® SKIN ist speziell entwickelt für die Sammlung von Hautproben für die Mikrobiom-Analyse. Das Kit ist geeignet für Hautproben aus trockenen, feuchten und talghaltigen Bereichen. Die effiziente Konservierungslösung stabilisiert die mikrobielle DNA effizient und verhindert so Verzerrungen des mikrobiellen Profils durch mikrobielles Wachstum oder DNA-Abbau. Die einfache Handhabung ist optimal für Eigenbeprobung durch Patienten.
Hard Facts .
- Ideal für Eigenbeprobung durch Patienten/Studienteilnehmer
- Geeignet für trockene, feuchte oder talgige Haut-Abnahmestellen
- Perfekte Konservierung von mikrobiellen Profilen (<30 Tage)
- Geeignet für viele Downstream-Applikationen (16S, ITS2 und WGS)
- Stabilisiert das mikrobielle Profil gegen Temperaturschwankungen (-20°C bis +50°C)
Anwendungen .
Das Swab-basierte OMNIgene®•SKIN (OMR-140) Kit ermöglicht einen perfekten Snapshot des humanen Haut-Mikrobioms. Auch von Hautpartien unterschiedlicher Beschaffenheit gelingt das Sammeln hochwertiger mikrobieller DNA.
Bouevitch, A., Macklaim, J., & Le François, B. (2020). OMNIgene®•SKIN (OMR-140): an optimized collection device for the capture and stabilization of the human skin microbiome. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00067.pdf
OMNIgene SKIN - OMR-140 .
Produktübersicht .
| Eigenschaft | OMNIgene SKIN (OMR-140) |
|---|---|
| Volumen der Stabilisierungslösung | 1 ml |
| Optimiert für Bakteriom- und Mykobiom-Profiling | ✔ |
| Stabilität bei Einfrieren/Auftauen und Temperaturschwankungen | -20ºC bis +50ºC |
| Validiert für 16S, ITS2 und metagenomische WGS-Sequenzierung | ✔ |
| Stabilität des Mikrobiomprofils bei Raumtemperatur | 30 Tage |
| Geeignet für alle Hauttypen (trocken, feucht, talgig) | ✔ |
| Validierte Methode, die Komponenten mit geringer biologischer Belastung enthält | ✔ |
| Integrierter 2D-Barcode für die Rückverfolgbarkeit der Proben und einen effizienten Arbeitsablauf | ✔ |

Vorteile .
- Selbstsammlung hochwertiger DNA von Hautstellen mit geringer mikrobieller Biomasse (trocken, feucht oder talgig)
- Stabilisierung von Bakterien- und Pilzprofilen am Ort der Entnahme von allen Hauttypen
- Die Probenstabilität von 30 Tagen bei Umgebungstemperatur eliminiert die Notwendigkeit einer sofortigen Lagerung im Gefrierschrank und einer Kühlkette beim Transport
- Realistische Darstellung der mikrobiellen Zusammensetzung durch Minimierung der durch mikrobielles Wachstum und DNA-Abbau verursachten Verzerrungen
- Isolierung qualitativ hochwertiger DNA, die sich für PCR, 16S, ITS2 und metagenomische Shotgun-Sequenzierung eignet
- Die intuitive Handhabung verbessert die Compliance der Spender und die Qualität der Probenennahme
- Perfekte Stabilisierung des mikrobiellen Profils gegenüber Temperaturschwankungen bei Versand- und Lagerung
- Ermöglicht nachgeschaltete Analyse auch bei geringer Besiedlungsdichte
Weitere Informationen .
-
HerstellerwebseiteHerstellerwebseite
-
Gebrauchsanweisung (engl.)Gebrauchsanweisung (engl.)
-
Data SheetData Sheet
Unverfälschte mikrobielle Profile .
Selbst nach 30-tägiger Lagerung bei Raumtemperatur zeigen sich keine signifikanten taxonomischen Veränderungen im mikrobiellen Profil der Probe.
Aus: Bouevitch, A., Macklaim, J., & Le François, B. (2020). OMNIgene®•SKIN (OMR-140): an optimized collection device for the capture and stabilization of the human skin microbiome. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00067.pdf
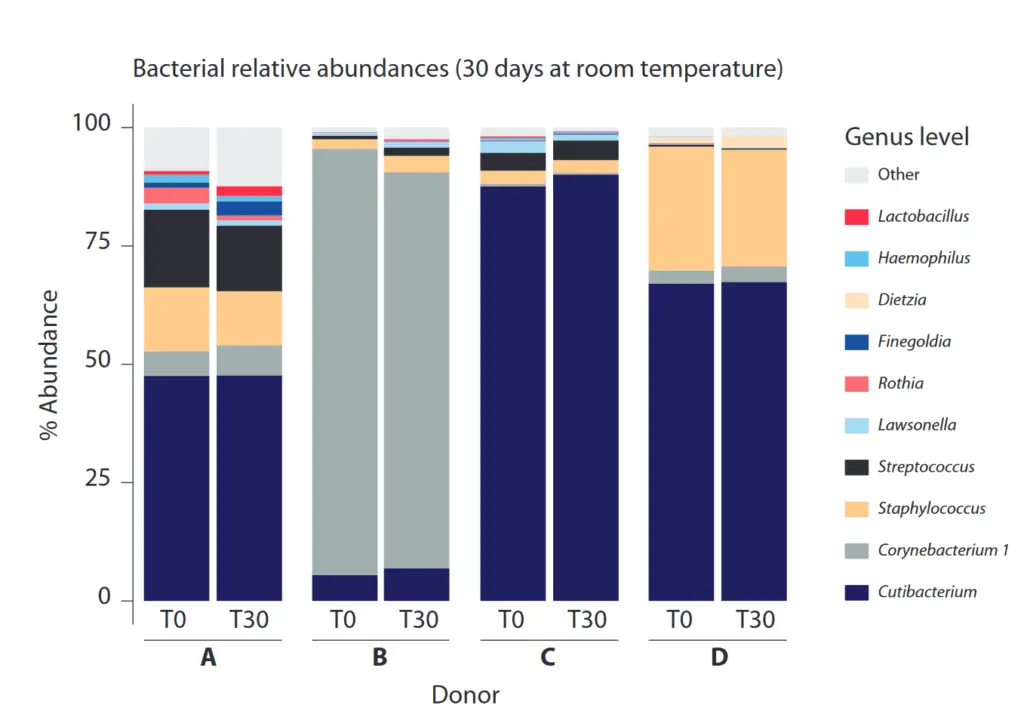
Stabilität des taxonomischen Profils in OMNIgene-SKIN-Proben, die bei Raumtemperatur für 30 Tage gelagert wurden. Das 16S rRNA-Gen aus T0- und T30-extrahierten Proben wurde mit Primern für V3-V4 amplifiziert, und das Diagramm der relativen Häufigkeit wurde aus den gefilterten Amplikon-Sequenzvarianten-Daten (ASV) erstellt, die mithilfe der SILVA-Datenbank klassifiziert und auf Gattungsebene oder die niedrigste zuordenbare taxonomische Ebene aggregiert wurden, wie in Klammern angegeben.
Taxonomische Profile von verschiedenen Probennahme-Stellen .
16S taxonomische Profile zeigten, dass OMNIgene-SKIN bakterielle Taxa erfasst, die bekanntermaßen mit bestimmten Hautstellen assoziiert sind (Abbildung Links). Zum Beispiel war Cutibacterium (formal bekannt als Propionibacterium) hauptsächlich mit mit talghaltigen Hautstellen (Gesicht und Kopfhaut) assoziiert, während Corynebacterium und Staphylococcus waren die beiden die beiden vorherrschenden Gattungen auf Zehenstegen (feuchte Hautstellen) waren. Auch die taxonomischen ITS2 Profile zeigten, dass Malassezia in hoher relativer Talg- und trockener Haut der meisten Spender in hoher relativer Häufigkeit Mehrheit der Spender gefunden wurde, während eine größere Vielfalt an Pilzgattungen eine größere Vielfalt an Pilzgattungen auf Zehenstegen festgestellt wurde (Abbildung Rechts).
Aus: Bouevitch, A., Macklaim, J., & Le François, B. (2020). OMNIgene®•SKIN (OMR-140): an optimized collection device for the capture and stabilization of the human skin microbiome. DNA Genotek. https://www.dnagenotek.com/ROW/pdf/PD-WP-00067.pdf

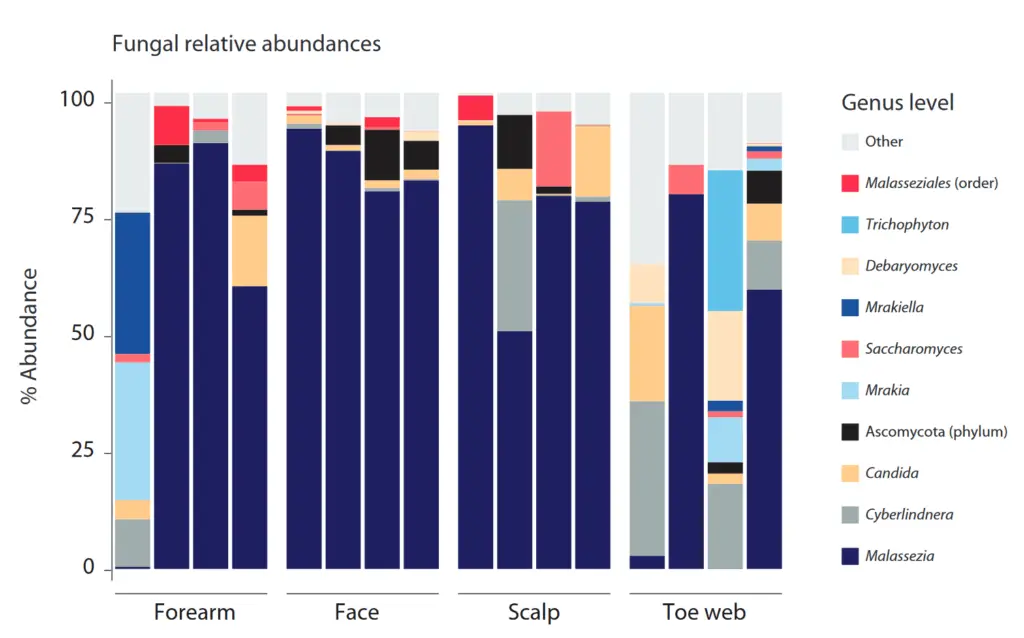
Bakterielle und pilzliche taxonomische Profile von mikrobiellen Hautproben, die am Unterarm (trocken), im Gesicht und auf der Kopfhaut (sebaceous) und Zehennägeln (nass) mit OMNIgene-SKIN.
LINKS: Das 16S rRNA-Gen wurde mit Primern für V3-V4 amplifiziert. Das Diagramm der relativen Häufigkeit wurde wurde aus den gefilterten Amplikon-Sequenzvarianten-Daten (ASV) erstellt, die mit der SILVA-Datenbank klassifiziert und auf Gattungsebene oder der niedrigsten oder die niedrigste zuordenbare taxonomische Ebene, wie in Klammern angegeben.
RECHTS: Für die Pilzanalyse wurde die ITS2-Region mittels PCR amplifiziert und die relative Häufigkeit wurde aus den gefilterten ASV-Daten erstellt, die mit der UNITE-Datenbank klassifiziert und auf Gattungsebene oder der niedrigsten zuordenbaren taxonomischen Ebene wie in Klammern angegeben. Sowohl in (A) als auch in (B) sind die 10 häufigsten Taxa von 4 repräsentativen Spendern dargestellt, während die übrigen Reads
gruppiert als “andere”.
Anweisungen zur Probenentnahme durch den Benutzer .
Schritt-für-Schritt Anweisung zur Selbstentnahme von Probenmaterial.

