

qPCR mit SYBR® Green .
Unser neuer primaQUANT CYBR ADVANCED qPCR Master Mix mit SYBR® Green wurde von Grund auf neu entwickelt und glänzt in allen wesentlichen Performance-Kriterien. Seine hohe Sensitivität (Detektionslimit 1 Template), breite Dynamic Range und hohe Robustheit garantieren reproduzierbare Ergebnisse, auch unter schwierigen Bedingungen.
Die primaQUANT CYBR ADVANCED Master Mixe sind sehr temperaturstabil. Sie müssen nicht auf Eis pipettiert werden und können problemlos mehrere Tage bei Raumtemperatur oder mehrere Wochen im Kühlschrank gelagert werden.
Die primaQUANT CYBR ADVANCED Master Mixe ersetzen die primaQUANT CYBR Master Mixe mit den Artikelnummern SL-9902, SL-9902R, SL-9902HR, SL-9902B, SL-9902RB, SL-9902HRB.
Hard Facts .
- Exzellente Sensitivität: 1 Template pro Reaktion
- Ultra-Fast: qPCR in 15 Minuten
- Stabil bei Raumtemperatur - Pipettieren ohne Eis
- Geeignet für Schmelzkurvenanalyse
- Volle Kontrolle: Wahlweise mit Blauem Farbstoff
- Made in Germany

Anwendungen .
Mit dem PrimaQUANT CYBR qPCR Master Mix können Sie Genotypsierungen mittels High-Resolution Melting Curves (HRM) durchführen – ganz ohne Agarose-Gel.
Li, W., Matsuoka, M., Kai, M., Thapa, P., Khadge, S., Hagge, D. A., Brennan, P. J., & Vissa, V. (2012). Real-Time PCR and High-Resolution Melt Analysis for Rapid Detection of Mycobacterium leprae Drug Resistance Mutations and Strain Types. Journal of Clinical Microbiology, 50(3), 742–753. https://doi.org/10.1128/JCM.05183-11
Słomka, M., Sobalska-Kwapis, M., Wachulec, M., Bartosz, G., & Strapagiel, D. (2017). High Resolution Melting (HRM) for High-Throughput Genotyping—Limitations and Caveats in Practical Case Studies. International Journal of Molecular Sciences, 18(11), 2316. https://doi.org/10.3390/ijms18112316
Lefever, S., Rihani, A., Van der Meulen, J., Pattyn, F., Van Maerken, T., Van Dorpe, J., Hellemans, J., & Vandesompele, J. (2019). Cost-effective and robust genotyping using double-mismatch allele-specific quantitative PCR. Scientific Reports, 9(1), 2150. https://doi.org/10.1038/s41598-019-38581-z
Die Genotypisierung von Single-Nukleotid-Polymorphismen (SNP) ist in genetischen Untersuchungen weit verbreitet, um die Faktoren zu bestimmen, die vererbten Merkmalen zugrunde liegen.
Baris, I., Etlik, O., Koksal, V., Ocak, Z., & Baris, S. T. (2013). SYBR green dye-based probe-free SNP genotyping: introduction of T-Plex real-time PCR assay. Analytical Biochemistry, 441(2), 225–231. https://doi.org/10.1016/j.ab.2013.07.007
Dhas, D. B. B., Ashmi, A. H., Bhat, B. V., Parija, S. C., & Banupriya, N. (2015). Modified low cost SNP genotyping technique using cycle threshold (Ct) & melting temperature (Tm) values in allele specific real-time PCR. The Indian Journal of Medical Research, 142(5), 555–562. https://doi.org/10.4103/0971-5916.171282
Pang, Y., Zhou, Y., Wang, S., Lu, J., Lu, B., He, G., Wang, L., & Zhao, Y. (2011). A novel method based on high resolution melting (HRM) analysis for MIRU-VNTR genotyping of Mycobacterium tuberculosis. Journal of Microbiological Methods, 86(3), 291–297. https://doi.org/10.1016/j.mimet.2011.05.016
Bai, L., Deng, Y.-M., Dodds, A. J., Milliken, S., Moore, J., & Ma, D. D. F. (2006). A SYBR green-based real-time PCR method for detection of haemopoietic chimerism in allogeneic haemopoietic stem cell transplant recipients. European Journal of Haematology, 77(5), 425–431. https://doi.org/10.1111/j.1600-0609.2006.00729.x
Weksberg, R., Hughes, S., Moldovan, L., Bassett, A. S., Chow, E. W., & Squire, J. A. (2005). A method for accurate detection of genomic microdeletions using real-time quantitative PCR. BMC Genomics, 6, 180. https://doi.org/10.1186/1471-2164-6-180
Zur Bestimmung der Expression eines Gens (Gen- oder RNA-Expression) muss zuvor die Gesamt-RNA in cDNA umgeschrieben werden.
Für die separate cDNA-Synthese ist unser primaREVERSE RT-Kit erste Wahl.
Alternativ können Sie auch unseren primaQUANT 1STEP One-Step RT-qPCR Kit verwenden und direkt RNA in einer qPCR einsetzen.
Die Länge der Telomere eignet sich gut als Biomarker für die Zellalterung. Damit lässt sich auch die Auswirkung von Umwelteinflüssen auf Prozesse der Zellalterung untersuchen. Die qPCR ist eine schnelle, zuverlässige und kostengünstige Methode zur Untersuchung von Telomerlängen.
Lin, J., Smith, D. L., Esteves, K., & Drury, S. (2019). Telomere length measurement by qPCR – Summary of critical factors and recommendations for assay design. Psychoneuroendocrinology, 99, 271–278. https://doi.org/10.1016/j.psyneuen.2018.10.005
Weitere Informationen:
Mit der Schmelzkurvenanalyse (High Resultion Melting Curve Analysis – HRM) lassen sich kleinste Änderungen im Genom wie z.B. SNP und Mutationen einfach mittels qPCR nachweisen.
Matsuda, K. (2017). PCR-Based Detection Methods for Single-Nucleotide Polymorphism or Mutation: Real-Time PCR and Its Substantial Contribution Toward Technological Refinement. Advances in Clinical Chemistry, 80, 45–72. https://doi.org/10.1016/bs.acc.2016.11.002
Simko, I. (2016). High-Resolution DNA Melting Analysis in Plant Research. Trends in Plant Science, 21(6), 528–537. https://doi.org/10.1016/j.tplants.2016.01.004
Mohammad Rahimi, H., Pourhosseingholi, M. A., Yadegar, A., Mirjalali, H., & Zali, M. R. (2019). High-resolution melt curve analysis: A real-time based multipurpose approach for diagnosis and epidemiological investigations of parasitic infections. Comparative Immunology, Microbiology and Infectious Diseases, 67, 101364. https://doi.org/10.1016/j.cimid.2019.101364
Next-Generation Sequencing erfordert für optimale Ergebnisse und hohe Ausbeuten eine genaue Quantifizierung der zu sequenzierenden DNA-Library.
Solche DNA-Libraries lassen sich kostengünstig, schnell und akurat mittels qPCR quantifizieren. Durch Vermeidung unnötig langer PCR Amplifikationen lässt sich der NGS Workflow optimieren, auch Amplifikationsverzerrungen werden minimiert.
Buehler, B., Hogrefe, H. H., Scott, G., Ravi, H., Pabón-Peña, C., O’Brien, S., Formosa, R., & Happe, S. (2010). Rapid quantification of DNA libraries for next-generation sequencing. Methods (San Diego, Calif.), 50(4), S15-18. https://doi.org/10.1016/j.ymeth.2010.01.004
Copy-Number Varianten können mittels SYBR-Green qPCR über unterschiedliche Cq Werte nach eine Kalibration gemessen werden. Zusätzlich erlaubt die Information der Schmelzkurve eine Abschätzung, ob es SNPs innerhalb der CNVs gibt.
Loewe, R. P. (2013). Combinational usage of next generation sequencing and qPCR for the analysis of tumor samples. Methods (San Diego, Calif.), 59(1), 126–131. https://doi.org/10.1016/j.ymeth.2012.11.002
Die Diagnostik von Pathogenen kann auch mittels Analyse von Nukleinsäuren durchgeführt werden.
DNA-Pathogene (DNA Viren, Bakterien, Pilze) können mittels quantitativer PCR direkt analysiert werden. Für RNA-Pathogene (RNA Viren, z.B. SARS-COV2) ist eine Umschreibung der RNA in cDNA notwendig. Hierfür bieten wir Ihnen mit dem primaREVERSE RT-Kit ein cDNA-Synthese Kit an. Alternativ können Sie mit unserem primaQUANT 1-STEP Master Mixe cDNA-Synthese und qPCR in einem Schritt durchführen.
Durch die besonders hohe Reinheit unserer primaQUANT Master Mixe können diese zur Qualitätskontrolle eingesetzt werden, um z.B. auszuschließen, dass ein Produkt mit Nukleinsäuren oder Nukleasen kontaminiert ist.
Features .
Hervorragende Performance .
Die primaQUANT ADVANCED Master Mixe bieten eine breite Dynamic Range und hervorragende Performance, auch bei geringen Templatemengen oder Targets mit niedriger Expression.
- Frühe Cq-Werte
- Breite Dynamic Range
- Niedrige Varianz innerhalb von Replikaten
- Limit-of-Detection von 1 Template pro Reaktion
- Nachweisgrenze bei cDNA von 0.1 pg je Reaktion
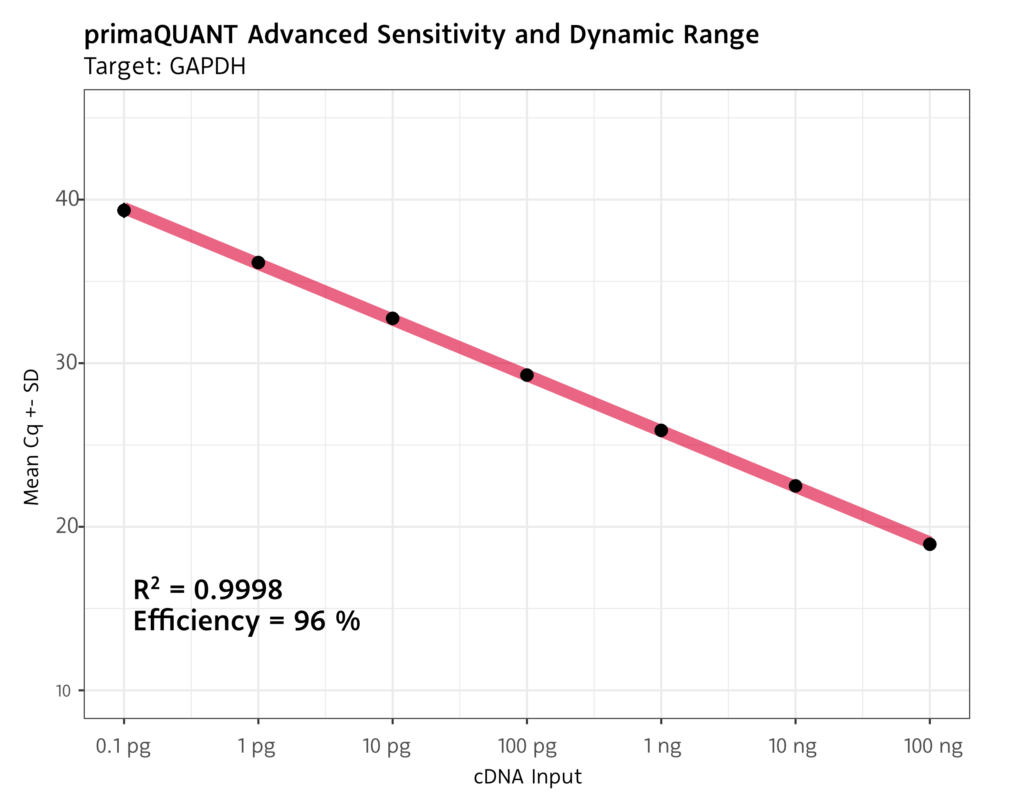
Robuste Quantifizierung selbst bei niedrigen Cq Werten am Detektionslimit.
Breite Dynamic Range mit exzellenter Effizienz.
Keine Templateinhibition bei großen Mengen an Input
Für alle qPCR Cycler .
Unsere qPCR 2x Master Mixe sind für alle gängigen qPCR-Cycler geeignet und wahlweise mit oder ohne ROX lieferbar.
Ob Ihr Gerät zur Normalisierung ROX (6-Carboxy- X-rhodamine) verwendet haben wir für Sie in einer Tabelle zusammengestellt.
| Hersteller | qPCR Gerät | ROX notwendig? |
|---|---|---|
| Applied Biosystems / Thermo Fischer | StepOne™ Real-Time PCR System | Ja (high ROX) |
| StepOnePlus™ Real-Time PCR System | Ja (high ROX) | |
| 7500 Real-Time PCR System | Ja | |
| 7500 Fast Real-Time PCR System | Ja | |
| 7500 Fast Dx Real-Time PCR Instrument | Ja | |
| 7500 Real-Time PCR System for Human Identification | Ja | |
| 7300 Real-Time PCR System | Ja (high ROX) | |
| Viia™ 7 Real-Time PCR System | Ja | |
| 7900HT Fast Real-Time PCR System | Ja (high ROX) | |
| OpenArray® Real-Time PCR Platform | Ja (high ROX) | |
| PRISM® 7000 Sequencing Detection System | Ja (high ROX) | |
| PRISM® 7700 Sequencing Detection System | Ja (high ROX) | |
| PRISM® 7900 Sequencing Detection System | Ja (high ROX) | |
| Gene Amp 5700 | Ja (high ROX) | |
| Quantstudio™ 3 / 3 FAST | Ja | |
| Quantstudio™ 5 / 5 FAST | Ja | |
| Quantstudio™ 6 | Ja | |
| Quantstudio™ 7 | Ja | |
| Quantstudio™ 12 | Ja | |
| QuantStudio™ 12K Flex system | Ja | |
| Bioneer | Exicycler™96 | Nein (optional) |
| Exicycler™384 | Nein (optional) | |
| Biorad | CFX96™ Real-Time PCR Detection System | Nein |
| CFX96 Touch™ Real-Time PCR Detection System | Nein | |
| CFX384 Touch™ Real-Time PCR Detection System | Nein | |
| CFX Opus 96 Real-Time PCR System | Nein | |
| CFX Opus 384 Real-Time PCR System | Nein | |
| CFX Connect™ Real-Time PCR Detection System | Nein | |
| iQ5 Real-Time PCR Detection System | Nein | |
| MiniOpticon™ Real-Time PCR Detection System | Nein | |
| Opticon 2 – Continuous Fluorescence Detection System | Nein | |
| Chromo4™ Four-Color Real-Time Detector | Nein | |
| Bioron | RealLine Cycler 48-4 | Nein (optional) |
| RealLine Cycler 48-5 | Nein (optional) | |
| RealLine Cycler 96-4 | Nein (optional) | |
| RealLine Cycler 96-5 | Nein (optional) | |
| DNA Technology | Dtlite | Nein (optional) |
| Dtprime | Nein (optional) | |
| Eppendorf | Mastercycler® ep realplex 4, Real-Time Thermal Cycler | Nein |
| Mastercycler® ep realplex 4s, Real-Time Thermal Cycler | Nein | |
| Mastercycler® ep realplex, Real-Time Thermal Cycler | Nein | |
| Mastercycler® ep realplex s, Real-Time Thermal Cycler | Nein | |
| Mastercycler Nexus | Nein | |
| Mastercycler Nexus gradient | Nein | |
| Mastercycler Pro | Nein | |
| Mastercycler Pro S | Nein | |
| Mastercycler Pro 384 | Nein | |
| Roche | LightCycler® 96 System | Nein |
| LightCycler® 480 System | Nein | |
| LightCycler® 2.0 Instrument | Nein | |
| LightCycler® 1.5 Instrument | Nein | |
| LightCycler® 1536 System | Nein | |
| LightCycler® Nano System | Nein | |
| Analytik Jena | qTower | Ja |
| qTower 2.0 | Ja | |
| qTower 2.2 | Ja | |
| qTower 3 | Ja | |
| qTower 3 84 | Ja | |
| Stratagene / Agilent | AriaMx Real-time PCR System | Nein |
| Mx3000P® qPCR System | Ja | |
| Mx3005P® qPCR System | Ja | |
| Mx4000® qPCR System | Ja | |
| Qiagen | Rotor-Gene™ Q | Ja |
| Rotor-Gene™ 6000 | Ja | |
| Quantabio | Q | Nein (optional) |
qPCR in unter 20 Minuten .
Extrem schnell
Die primaQUANT qPCR Master Mixe kommen wegen ihrer ultraschnellen DNA-Polymerase mit kürzesten Zyklenzeiten zurecht: Ein kompletter qPCR-Lauf dauert unter geeigneten Bedingungen weniger als 20 Minuten.
Selbstverständlich können Sie auch Ihr bisheriges qPCR Protokoll nutzen.
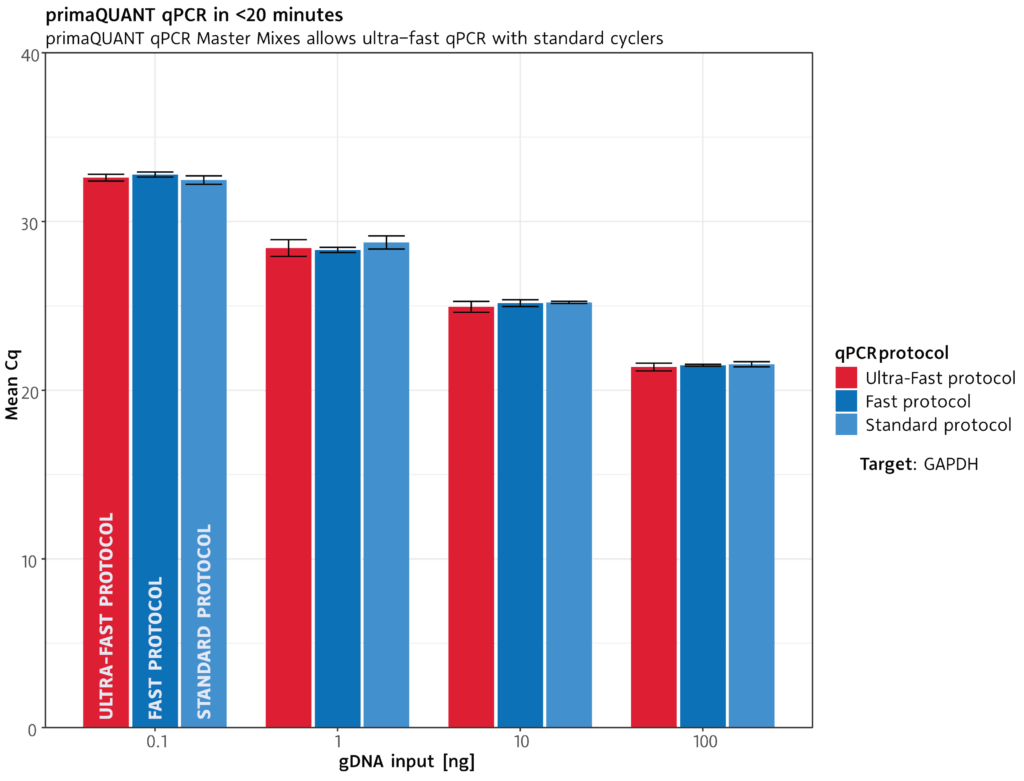
Standard-Protokoll:
| Schritt | Zeit | Temperatur | |
|---|---|---|---|
| Initiale Denaturierung | 1-3 Minuten | 92°C – 95°C | |
| 25 – 40 Zyklen | Denaturierung | 5-10 Sekunden | 92°C – 95°C |
| Kombiniertes Annealing/Elongation | 10-20 Sekunden | 60°C – abhängig von den verwendeten Primern |
Fast-Protokoll:
| Schritt | Zeit | Temperatur | |
|---|---|---|---|
| Initiale Denaturierung | 1 Minute | 92°C – 95°C | |
| 25 – 40 Zyklen | Denaturierung | 5 Sekunden | 92°C – 95°C |
| Kombiniertes Annealing/Elongation | 10 Sekunden | 60°C – abhängig von den verwendeten Primern |
Ultra-Fast-Protokoll:
Immer stabil .
Einfrieren/Auftauen
Unseren PCR- und qPCR-Reagenzien kann auch mehrfaches Einfrieren/Auftauen nichts anhaben. Sie können die Mixe daher problemlos mehr als 10x Einfrieren und wieder auftauen.
Lagerung im Kühlschrank
Die primaQUANT Master Mixe sind über Wochen bei 4°C stabil. Für kurzzeitige Lagerung empfehlen wir generell die Aufbewahrung bei 4°C im Kühlschrank.
Stabilität bei Raumtemperatur
Die primaQUANT Master Mixe wurden so optimiert, dass sie sogar mehrere Tage ohne Kühlung schadlos überstehen. Sie müssen Ihre qPCR daher nicht mehr aufwändig auf Eis pipettieren, um einen Sensitivitätsverlust zu vermeiden!


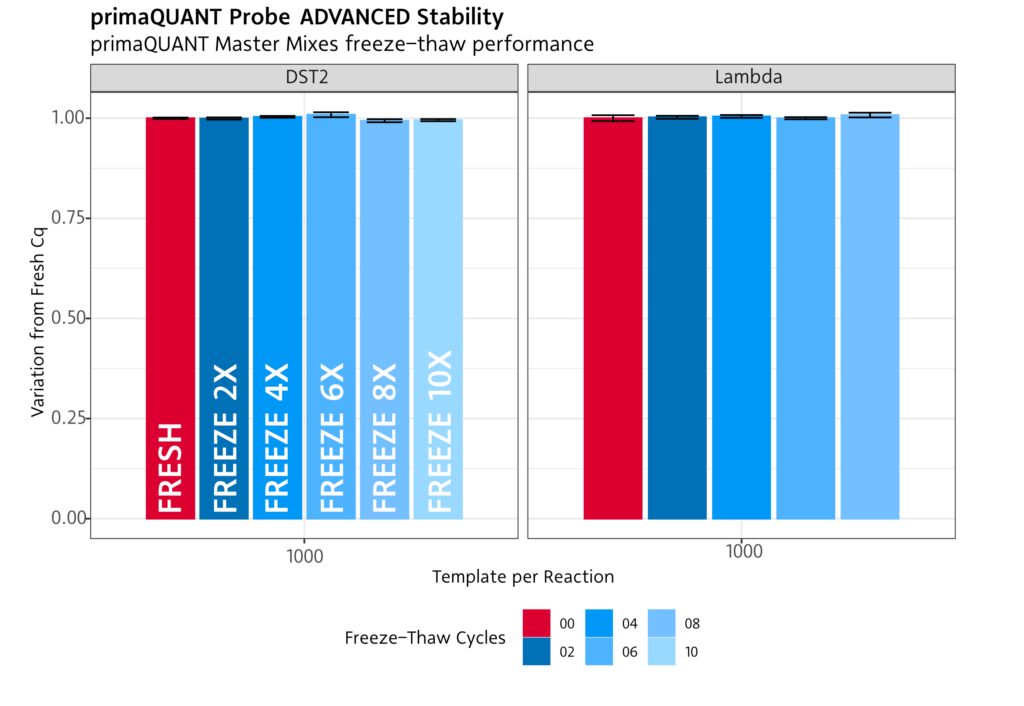
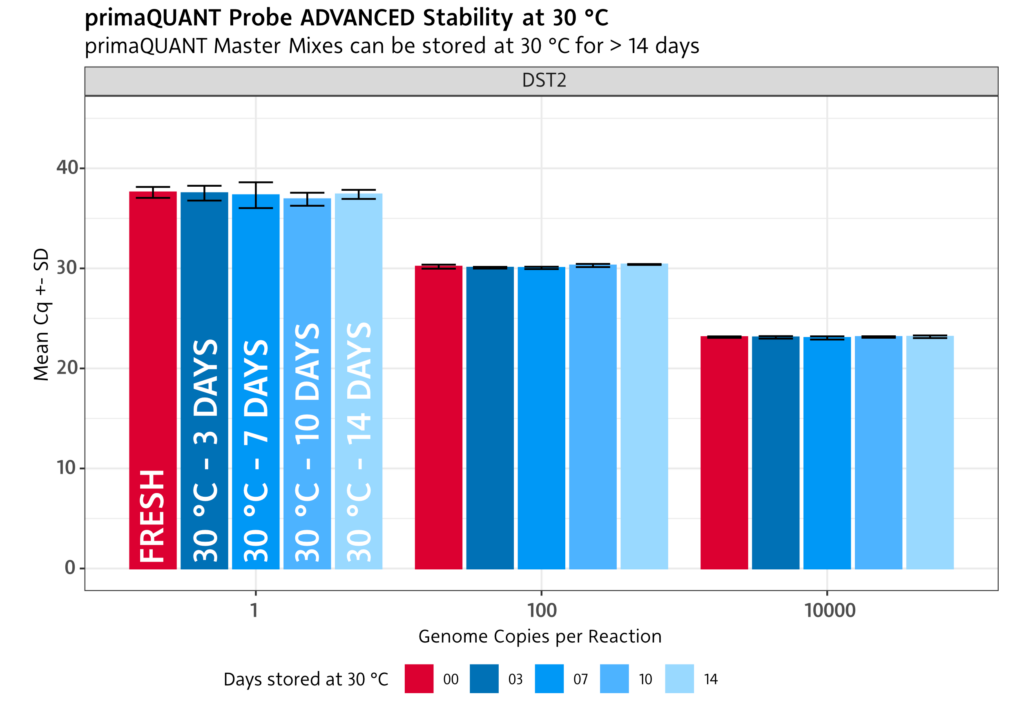
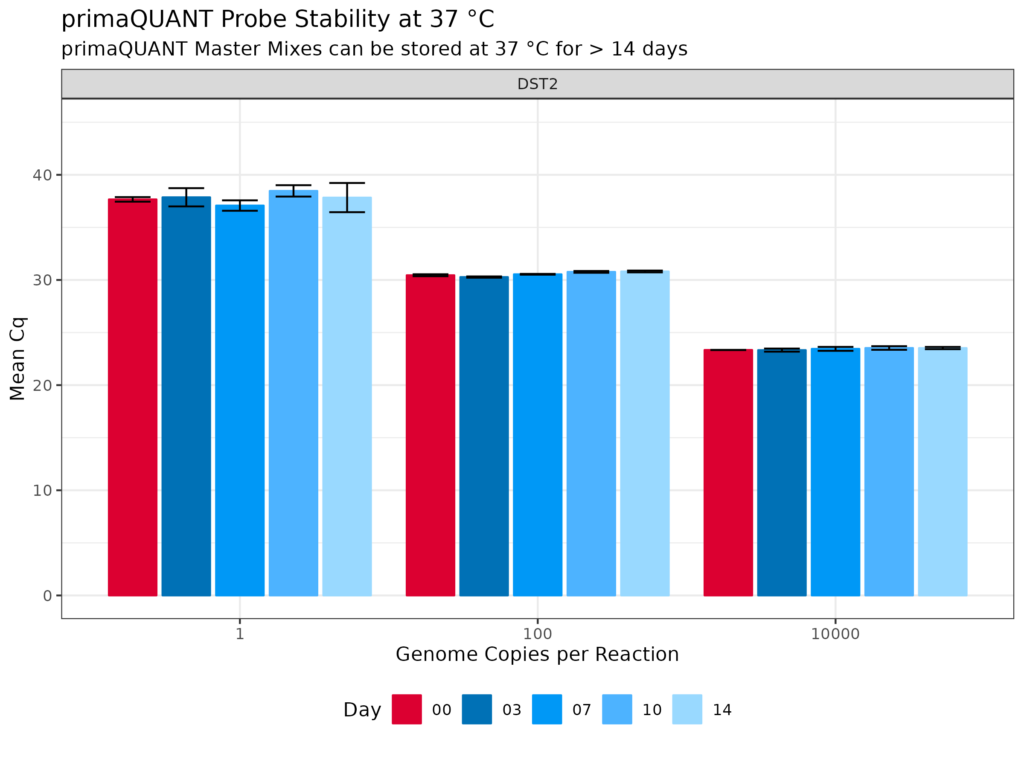
Praktische Abfüllung .
Kein lästiges Aliquotieren mehr!
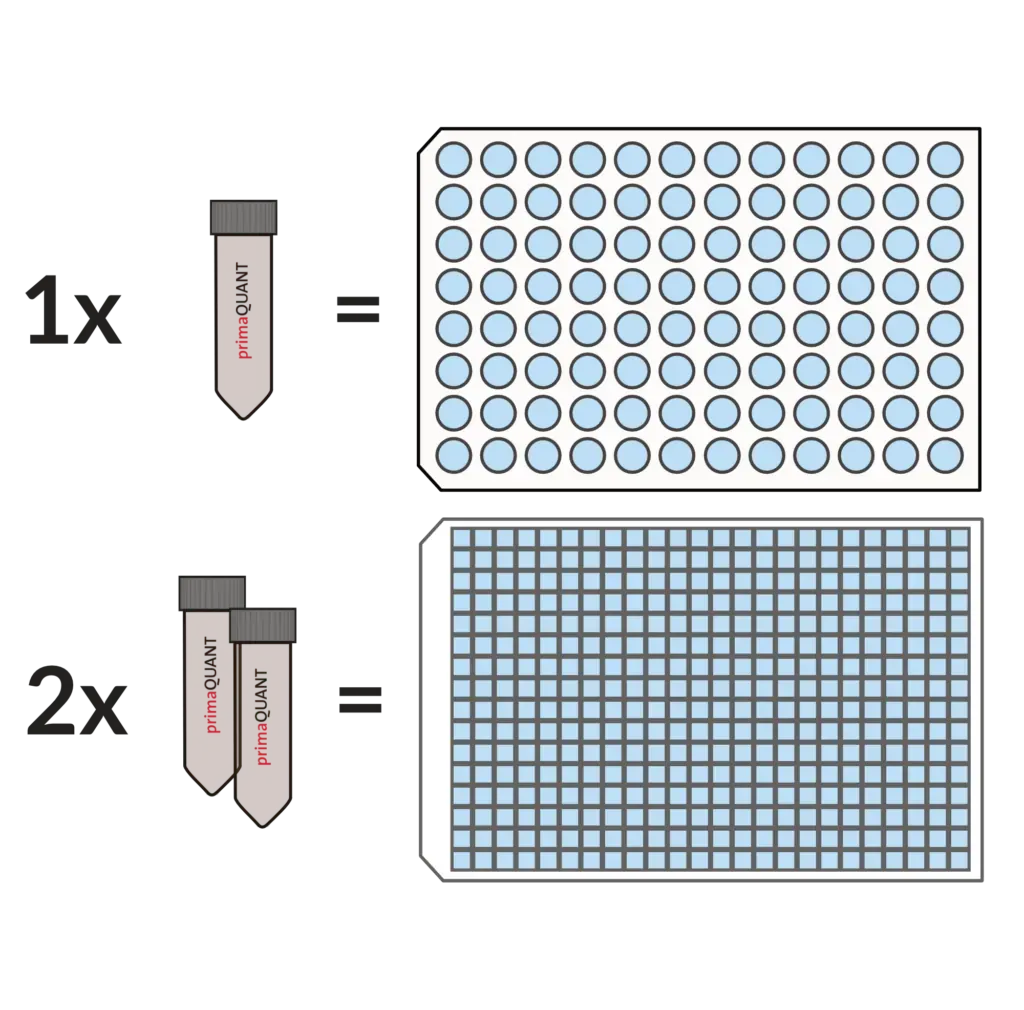
Unsere Master Mixe kommen standardmäßig in Einheiten von 1 ml.
Das ist sehr komfortabel und praktisch, denn ein Tube reicht für genau eine 96er Platte, zwei Tubes für eine 384er Platte. Sollten Sie einmal weniger benötigen, können Sie die Mixe einfach im Kühlschrank über mehrere Wochen lagern.
Kundenspezifische Abfüllungen: Als Hersteller können wir Ihnen selbstverständlich bei größeren Bestellungen auch Abfüllungen und Mixturen entsprechend Ihren speziellen Erfordernissen liefern.
Made in Germany .
Unsere Enzyme und Master Mixe entwickeln und produzieren wir ausschließlich selbst in Deutschland. Davon profitieren Sie gleich mehrfach:
- Sehr kurze Lieferzeiten
- Schneller und kompetenter Support
- ISO-zertifizierte Produktion
- High-end Qualitätskontrolle
- Kundenspezifische Abfüllungen oder Produktanpassungen

ADVANCED SYBR® Green Master Mixe .
Erhältlich mit ROX oder ohne ROX – passend für alle qPCR Geräte.
Downloads und weitere Informationen .
| Hersteller | qPCR Gerät | ROX notwendig? |
|---|---|---|
| Applied Biosystems / Thermo Fischer | StepOne™ Real-Time PCR System | Ja (high ROX) |
| StepOnePlus™ Real-Time PCR System | Ja (high ROX) | |
| 7500 Real-Time PCR System | Ja | |
| 7500 Fast Real-Time PCR System | Ja | |
| 7500 Fast Dx Real-Time PCR Instrument | Ja | |
| 7500 Real-Time PCR System for Human Identification | Ja | |
| 7300 Real-Time PCR System | Ja (high ROX) | |
| Viia™ 7 Real-Time PCR System | Ja | |
| 7900HT Fast Real-Time PCR System | Ja (high ROX) | |
| OpenArray® Real-Time PCR Platform | Ja (high ROX) | |
| PRISM® 7000 Sequencing Detection System | Ja (high ROX) | |
| PRISM® 7700 Sequencing Detection System | Ja (high ROX) | |
| PRISM® 7900 Sequencing Detection System | Ja (high ROX) | |
| Gene Amp 5700 | Ja (high ROX) | |
| Quantstudio™ 3 / 3 FAST | Ja | |
| Quantstudio™ 5 / 5 FAST | Ja | |
| Quantstudio™ 6 | Ja | |
| Quantstudio™ 7 | Ja | |
| Quantstudio™ 12 | Ja | |
| QuantStudio™ 12K Flex system | Ja | |
| Bioneer | Exicycler™96 | Nein (optional) |
| Exicycler™384 | Nein (optional) | |
| Biorad | CFX96™ Real-Time PCR Detection System | Nein |
| CFX96 Touch™ Real-Time PCR Detection System | Nein | |
| CFX384 Touch™ Real-Time PCR Detection System | Nein | |
| CFX Opus 96 Real-Time PCR System | Nein | |
| CFX Opus 384 Real-Time PCR System | Nein | |
| CFX Connect™ Real-Time PCR Detection System | Nein | |
| iQ5 Real-Time PCR Detection System | Nein | |
| MiniOpticon™ Real-Time PCR Detection System | Nein | |
| Opticon 2 – Continuous Fluorescence Detection System | Nein | |
| Chromo4™ Four-Color Real-Time Detector | Nein | |
| Bioron | RealLine Cycler 48-4 | Nein (optional) |
| RealLine Cycler 48-5 | Nein (optional) | |
| RealLine Cycler 96-4 | Nein (optional) | |
| RealLine Cycler 96-5 | Nein (optional) | |
| DNA Technology | Dtlite | Nein (optional) |
| Dtprime | Nein (optional) | |
| Eppendorf | Mastercycler® ep realplex 4, Real-Time Thermal Cycler | Nein |
| Mastercycler® ep realplex 4s, Real-Time Thermal Cycler | Nein | |
| Mastercycler® ep realplex, Real-Time Thermal Cycler | Nein | |
| Mastercycler® ep realplex s, Real-Time Thermal Cycler | Nein | |
| Mastercycler Nexus | Nein | |
| Mastercycler Nexus gradient | Nein | |
| Mastercycler Pro | Nein | |
| Mastercycler Pro S | Nein | |
| Mastercycler Pro 384 | Nein | |
| Roche | LightCycler® 96 System | Nein |
| LightCycler® 480 System | Nein | |
| LightCycler® 2.0 Instrument | Nein | |
| LightCycler® 1.5 Instrument | Nein | |
| LightCycler® 1536 System | Nein | |
| LightCycler® Nano System | Nein | |
| Analytik Jena | qTower | Ja |
| qTower 2.0 | Ja | |
| qTower 2.2 | Ja | |
| qTower 3 | Ja | |
| qTower 3 84 | Ja | |
| Stratagene / Agilent | AriaMx Real-time PCR System | Nein |
| Mx3000P® qPCR System | Ja | |
| Mx3005P® qPCR System | Ja | |
| Mx4000® qPCR System | Ja | |
| Qiagen | Rotor-Gene™ Q | Ja |
| Rotor-Gene™ 6000 | Ja | |
| Quantabio | Q | Nein (optional) |
Zusammensetzung
| Komponente | Stock Konzentration | 20 µl Reaktion | 10 µl Reaktion | Finale Konzentration |
|---|---|---|---|---|
| primaQUANT CYBR Master Mix | 2x | 10 µl | 5 µl | 1 x |
| Reverse Primer | 4 µM | 1 µl | 0,5 µl | 200 nM (100 – 400 nM empfohlen) |
| Forward Primer | 4 µM | 1 µl | 0,5 µl | 200 nM (100 – 400 nM empfohlen) |
| Template (DNA/cDNA) | – | variabel | variabel | 0,1 – 10 ng/Reaktion |
| Steriles Wasser | – | auf 20 µl | auf 10 µl | – |
Standard Protokoll
3-Step
| Schritt | Zeit | Temperatur | |
|---|---|---|---|
| Initiale Denaturierung | 1-3 Minuten | 92°C – 95°C | |
| 25 – 40 Zyklen | Denaturierung | 5-10 Sekunden | 92°C – 95°C |
| Annealing | 1-5 Sekunden | 60°C – abhängig von den verwendeten Primern | |
| Extension | 10-20 Sekunden | 72°C |
2-Step
| Schritt | Zeit | Temperatur | |
|---|---|---|---|
| Initiale Denaturierung | 1-3 Minuten | 92°C – 95°C | |
| 25 – 40 Zyklen | Denaturierung | 5-10 Sekunden | 92°C – 95°C |
| Kombiniertes Annealing/Elongation | 10-20 Sekunden | 60°C – abhängig von den verwendeten Primern |
Ultra-FAST Protokoll
3-Step
| Schritt | Zeit | Temperatur | |
|---|---|---|---|
| Initiale Denaturierung | 1 Minute | 92°C – 95°C | |
| 25 – 40 Zyklen | Denaturierung | 1-5 Sekunden | 92°C – 95°C |
| Annealing | 1-5 Sekunden | 60°C – abhängig von den verwendeten Primern | |
| Extension | 1 Sekunde | 72°C |



